扩增子测序
- 产品介绍
- 案例1
- 案例2
- 案例3
- 注意事项
微生物群落多样性分析是基于第二代高通量测序平台,对特定长度的 PCR 产物或者捕获的片段进行测序,例如16S/18S/ITS 等序列。然后分析相应环境下微生物群落的多样性和分布规律。

环境微生物多样性分析技术流程图
分析内容
1. 原始数据除杂及质量评估
2. OTU 聚类
3. 稀释曲线
4. 丰度分布曲线
5. 单样品 Alpha 多样性分析
6. 物种丰度差异分析
7. 多样品相似度比对
8. 根据客户实际需要进行个性化分析
样品信息
1. 样品类型: DNA;
2. 样品需求量:≥ 500 ng 样品质量要求 OD 260/280 应在 1.8 到 2.0 之间;
3. 电泳检测无明显 RNA 条带,基因组条带清晰、完整。所送的样品不要有多糖或糖蛋白污染;
4. 样品保存期间切忌反复冻融,送样时请使用冰袋或干冰运输。
肠癌演变不同阶段的肠道粘膜微生物
背景 (Background)
肠癌为结肠癌和直肠癌的总称,是胃肠道中常见的恶性肿瘤。肠癌的演变是从正常肠道粘膜演变为息肉,然后进一步癌化。已有研究表明,肠道微生物与肠癌的演变有很大关系。但是目前并不知道在肠癌演变的过程中微生物群落时如何变化的,哪些微生物在肠道从正常演变为腺瘤再到癌变过程中起了作用。因此想要确定在肠癌演变中不同阶段肠道微生物的组成。
结果 (Results)
研究主要思路是通过对正常肠道粘膜,腺瘤性息肉和癌变了的肠道粘膜上微生物进行16S rRNA 测序分析,确定不同演变阶段的肠道微生物群体,观察这些群体与癌变状态的关系以及微生物之间的关系。总共收集了 160 个人的肠道粘膜样本。160 个人包括 61 个正常人,47 个有肠道息肉的和 52 个确诊为肠癌的。采样位置为正常样本的肠道粘膜,长有息肉的肠道。对息肉和息肉旁的肠道粘膜进行了取样,同样对肠癌的肠道是对癌细胞和癌旁的肠道粘膜进行了取样。主要分析了正常肠道,腺瘤肠道和癌变肠道中病灶和病灶旁肠道粘膜上的微生物的物种组成。对这些微生物的聚类得到了5 类 metacommunity,而其中一类 metacommunity 主要是口腔微生物,并且主要来自肠癌样本。对病灶和病灶旁成对样本的分析表明病灶和病灶旁微生物存在着显著的差异,并给出了不同演变阶段存在显著差异的微生物;在病变样本中微生物会出现生态失调,伴随着出现微生物的互斥关系;并且找到了与癌变发展密切相关的一些微生物群体。
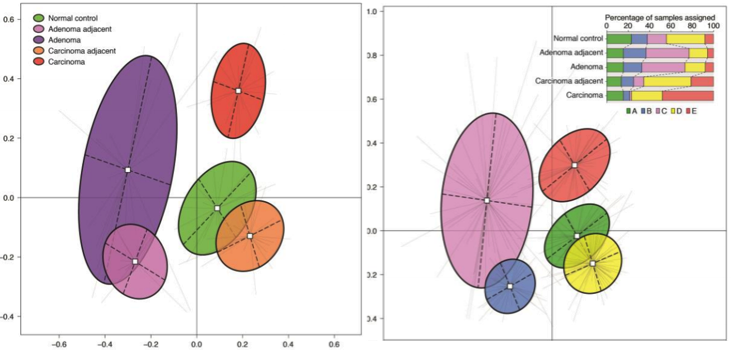
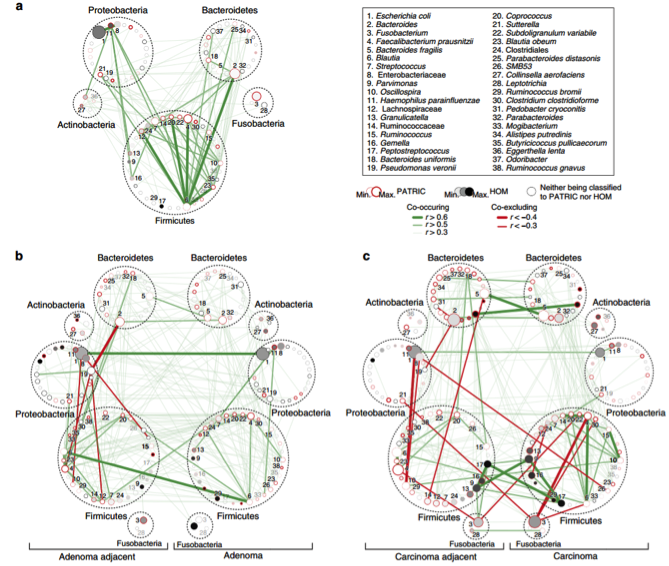
原文: Nakatsu, G. et al. Gut mucosal microbiome across stages of colorectal carcinogenesis. Nature Communications, 6(2015).
人类基因组塑造肠道微生物
背景 (Background)
随着人们对身材的愈加重视,已经有了很多关于代谢疾病和肥胖的研究。已有研究证据提示肠道细菌是人体不可或缺的一部分,肠道菌与代谢疾病和肥胖症有着密切联系。而且人们普遍认为遗传变异与代谢性疾病及胖瘦之间也存在相关性。然而,人们并不清楚遗传变异与肠道菌的组成及丰度之间的关系。在以前的研究中,人类遗传变异和肠道菌群多样性之间的关系被假定为可忽略不计。根据之前的研究,发现家庭成员之间因为受相同的环境因子的影响而具有更相似的微生物。而亲属之间有很高的遗传一致性,由此我们猜测相同的基因组成可能是家庭成员之间微生物更相似的基础条件。基于之前的先验知识与已有的研究成果,本文开展基因组与肠道微生物之间的关系研究。本文主要回答以下三个问题:第一,肠道微生物中哪些种类是可遗传的;第二,预测到的宏基因组功能,哪些是可以遗传的;第三,可遗传的微生物是如何对宿主的体重产生关系的。最后,通过微生物移植的方法来观察,遗传性的微生物对表型的影响。
结果 (Results)
研究人员收集了416 对双胞胎的 1 千份粪便样本,并对其中的微生物进行了16S rRNA 基因测序。与异卵双胞胎相比,同卵双胞胎之间的微生物丰度更为相似。异卵双胞胎的基因相似性其实跟普通兄妹差不多,只不过他们在子宫里共享了同样的环境。这样的结果说明,基因对肠道菌的组成有着深远的影响。研究人员发现,受宿主遗传学背景影响的是细菌Christensenellaceae。这种细菌对人体健康有益,在身材较瘦的人体内丰度更高。研究人员用这种细菌处理小鼠,抑制了这些小鼠的体重增加。由此可见,提高这种细菌的丰度,有望帮助人们避免肥胖或者有效减肥。
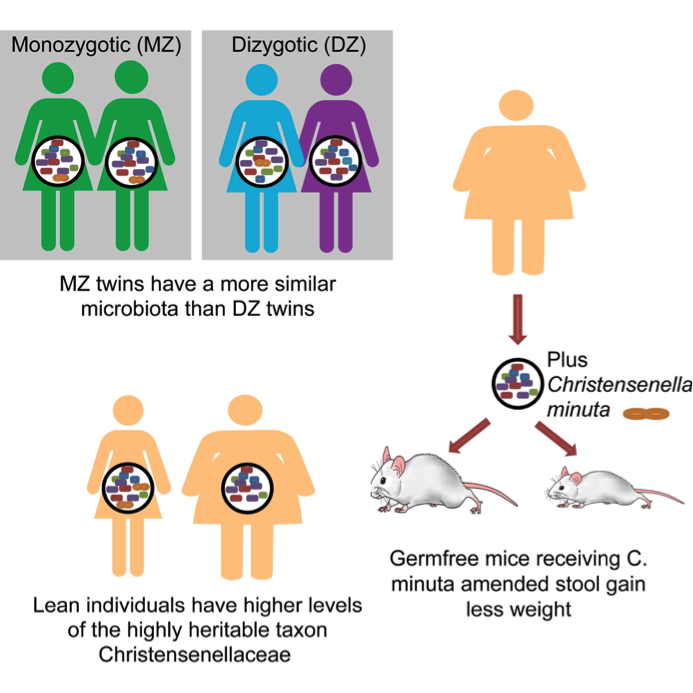
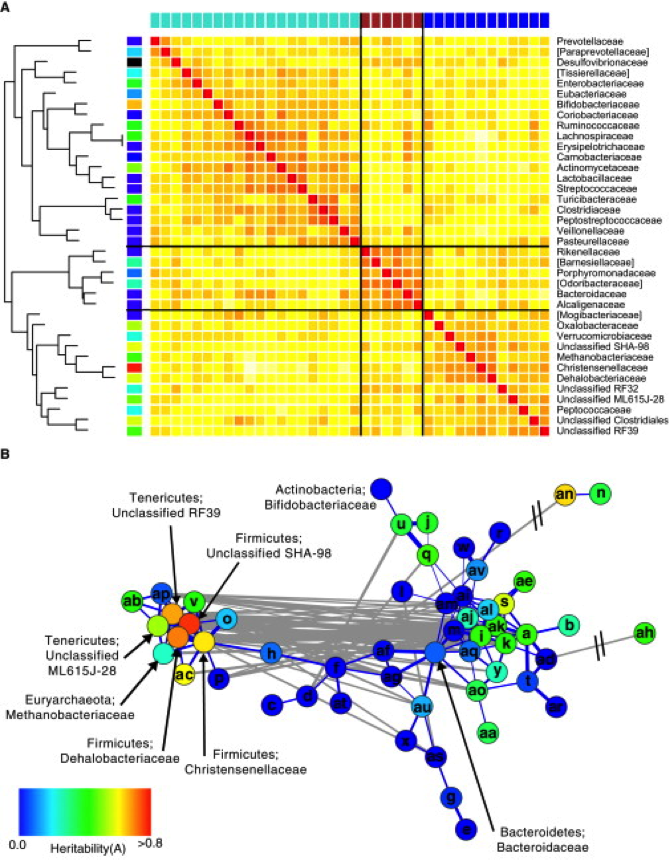
原文: Goodrich, J. K. et al. Human genetics shape the gut microbiome.Cell, 159:789-99(2014).
海洋微生物的多样性
背景 (Background)
为了研究全球变暖对海洋生物多样性的影响,联合国环境署 (UNEP) 斥巨资,由 Tara Ocaens 领导的研究团队横跨大西洋、太平洋、南极和印度洋7万英里海域,结合生态学、系统生物学和海洋学,对海洋微生物进行采样来探究浮游生物在地球生命系统中的角色、气候变化对海洋食物链的影响等。历时三年半的时间从 (2009年11月份至2013年5月份),从210个点位获取了3万5千个海洋样本,通过对环境因子的监测和样本中16S rRNA和18S rRNA扩增子的测序及分析,获得了一系列的重大发现。
结果 (Results)
首先这项研究得到了很多珍贵的数据资源:比如海洋微生物基因列表、浮游生物包括病毒、原核生物、微小真核生物的多样性普查表等。在真核生物方面,总共发现了15万基因不同的真核生物,其中很多是单细胞生物;很多浮游生物存在着寄生关系,这能够将营养物质回收到食物链的底层。该研究还发现了多于5000种的病毒群落,其中只有39种与已知的病毒相似,此外,发现在阳光能够触及的深度,温度是影响微生物群落结构的主要因素,也就是说不同生物根据海水的温度聚集在一起。研究人员还分析了海洋病毒的全球分布,发现病毒具有很高的局部多样性和较低的全局多样性,验证了一个十年前提出的理论:病毒是在局部“种子库”产生的,它们顺着洋流漂浮,最终不同的地区有不同的病毒群体。TaraOcean团队对海洋进行了系统性的取样,涵盖了从病毒到动物所有的生物域,收集了丰富多样的环境数据。这项研究奠定的基础,有助于评估气候对海洋生态系统的影响。
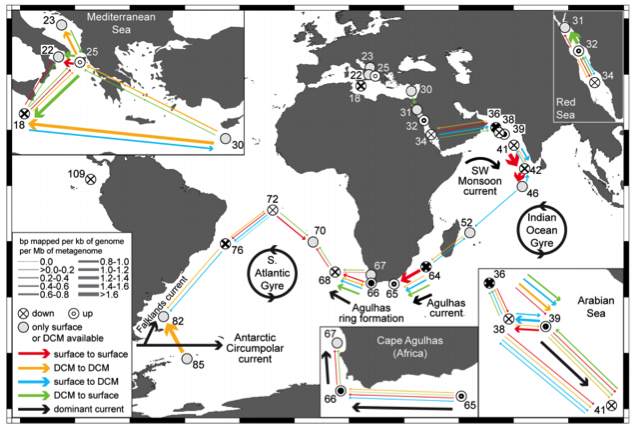

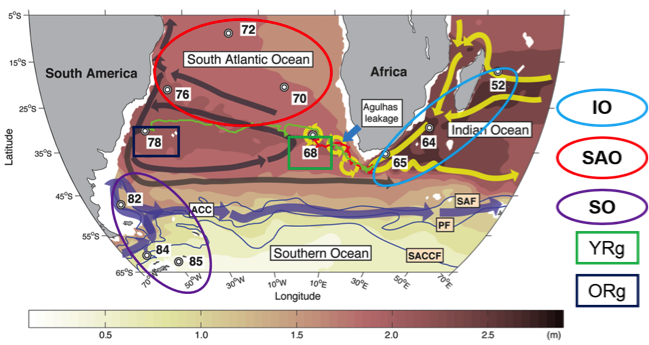
原文: Marine ecology: Ocean survey finds huge diversity. Nature, 2015, 521.
1. Lima-Mendez, G. et al. Determinants of community structure in the global plankton interactome.Science 348, doi:10.1126/Science.1262073 (2015).
2. de Vargas, C. et al. Eukaryotic plankton diversity in the sunlit ocean. Science348,doi:10.1126/Science.1261605 (2015).
3. Brum, J. R. et al. Patterns and ecological drivers of ocean viral communities. Science 348,doi:10.1126/Science.1261498 (2015).
4. Sunagawa, S. et al. Structure and function of the global ocean microbiome. Science 348,doi:10.1126/Science.1261359 (2015).
5. Villar, E. et al. Environmental characteristics of Agulhas rings affect interoceanplankton transport.Science 348, doi:10.1126/Science.1261447 (2015).
1. 哪些环境样品可以进行微生物多样性检测
针对样品如人体口腔、胃肠道; 动物的瘤胃、肠道; 植物的菌根进行研究;针对环境相关样品,如土壤 (草原和矿地)、水体(海水和河水)、空气等进行研究。
2. 分子指纹技术与高通量测序技术在环境微生物群落多样性研究中有什么区别
DGGE 等分子指纹技术,在其实验结果中往往只含有数十条条带,只能反映出样品中的优势种群,也无法得到细菌种类的数量。而高通量测序技术能同时对样品中的优势种群及微量的劣势种群进行检测,获得样品中的微生物群落组成,并将其含量进行数字化。
3. 于传统研究技术相比,基于高通量测序在环境微生物多样性检测技术有何优势
1) 常规的宏基因组学研究方法包括基因克隆文库、变性梯度凝胶电泳 DGGE/TGGE 等,但这些方法的通病是信息量太小,不能充分反映复杂的环境微生物多样性和分布。
2) 基因克隆文库构建和检测的工作量大,且自然界中 99% 的微生物在实验室都没有办法纯化培养,从培养基上挑取克隆菌株,摇菌转化测序,效率低下。DGGE 法曾经广泛应用于检测微生物群落结构的多态性,但是需要标准菌株,且受到凝胶电泳特性的局限,无法检测到稀有菌群的种类,因此其重复性和分辨率都不甚理想。
3)第二代高通量测序无需构建质粒克隆文库,这避免了文库构建过程中利用宿主菌对样品进行克隆而引起的系统偏差,可以直接对环境样品中的基因组片段进行测序,简化了基本操作,提高了测序效率,它能够对一个群落中微生物的多样性作更加深入和全面的描述,且具有通量高,重复性好,准确度高的优点,因而在微生物生态学研究中逐渐占据了优势。
