Microbiome: 环境样品中微生物群落物种丰度绝对定量生信分析方法的实现
在《Absolute quantitation of microbiota abundance in environmental samples》一文中, 作者使用生信分析方法实现环境样品中微生物群落丰度的绝对定量。
文章中生物信息分析主要包括扩增子分析、多维标度分析和T检验分析。
1)扩增子分析采用Usearch10,且采用Unoise3非聚类直接去噪生成ZOTUs(Zero-radius OTUs),但该软件的64位版本收费,而免费的32位版对数据分析量有限制。
2)采用PRIMER6软件进行降维方法非度量多维标度 (Non-metric Multidimensional scaling,NMDS)来分析两组微生物群落结构组成是否存在显著差异。
3)采用统计学方法中的T检验并进行Bonferroni 矫正来检验两组样品之间物种组成是否存在显著性差异。
扩增子分析
分析步骤如下:
1.扩增子分析采用Usearch 10,且查找OTU使用非聚类算法unoise3(我们展示最新版本Usearch 11操作方法);
2.三类扩增子注释数据库分别采用SILVA, PR2 and ITSone数据库;
3.扩增子分析时,需先将PEF三种合成spikes的序列和注释信息分别加入到各自数据库中;
4. 最终得到OTU代表序列需满足各自的限定长度。
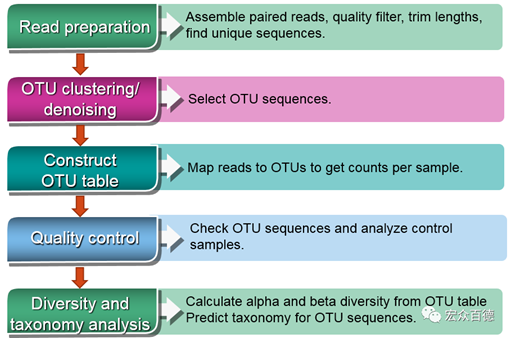
Step1:Merge paired reads(双端测序数据合并)
Step2:Strip primers (去除前后端引物片段)
Step3:Quality filter(质控)
Step4:Find unique read sequences and abundances (去冗余)
Step5:Denoise: predict biological sequences and filter chimeras(查找ZOTUs&去嵌合体)
Step6:Make OTU table & Normalize to 5k reads for each sample(生成OTU table表并进行均一化)
Step7:Predict taxonomy(对每个OTU进行物种注释)
Step8:Taxonomy summary reports(生成不同水平下的物种注释汇总表)
上述Usearch11的8个步骤已满足本文献中所有扩增子分析需求,后续的分析均以Step6或Step8得到的Table表为基础,通过格式转化统计相应基因的拷贝数,或通过比较合成spikes和某物种的相对丰度并结合加入spikes的实际含量,计算得出对应物种绝对含量等,最后实现可视化(点图/折线图/柱状图等可利用R实现)及美化(可采用Adobe Illustrator CS6软件向结果图添加其他元素)。
NMDS分析
分析步骤如下:
1.软件下载地址:https://www.primer-e.com/download/,安装软件,进入操作界面
2.点击File上传ZOTUs Table表,txt文件即可,选择Data type,勾选基本属性,导入物种丰度表
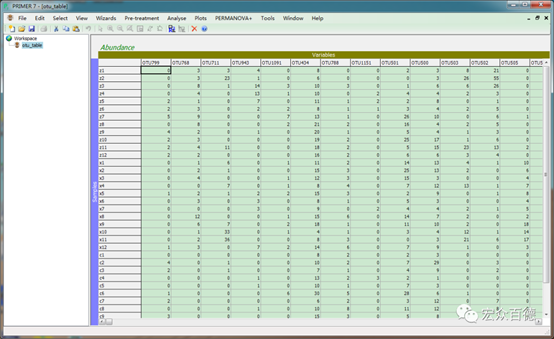
3.点击Pre-treatment→Transform(Overall)进行数据预处理,格式转化,文章采用的是取平方根Square root
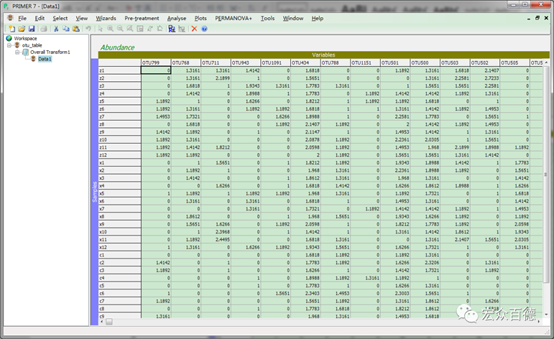
4.点击Analyse→Resemblance计算样品间的距离矩阵,方法选择Bray-Curtis similarity
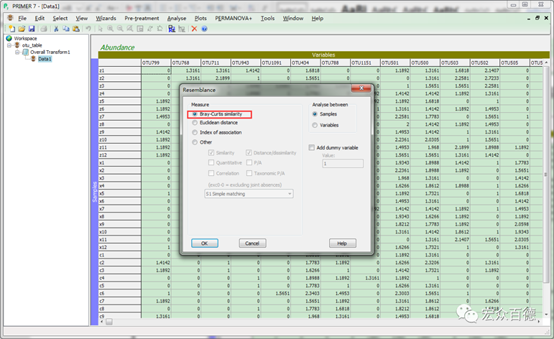
5.点击Analyse→MDS→Non-metric MDS(nMDS)…,进行非度量多维标度(NMDS)分析
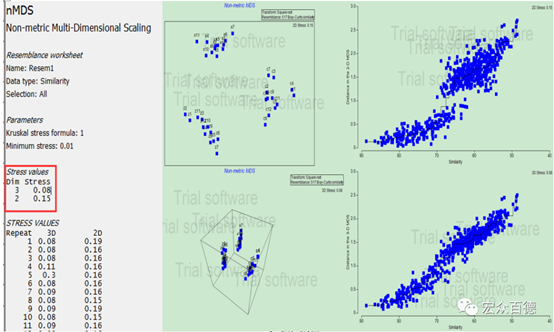
非度量多维标度(NMDS)分析参数设置:建议参数选择如下,其中Number of restarts迭代次数较重要,直接影响结果的可靠性,默认值为50,操作文档建议设置为100。结果展示如下,样品间的距离越近,表示其物种组成越接近。
T检验
为了比较两种土壤特定物种的相对/绝对丰度是否存在显著差异,文章采用了T检验法并进行Bonferroni多重检验矫正,将最终矫正得到的p_value值(为了区分用q表示)作为是否存在显著差异的评判标准,在文章中“*”表示q< 0.05, “***”表示q < 0.01, “***”表示q < 0.001。
T检验的R语言分析流程:
Step1:安装并载入分析需要的包
Step2:读入文件
Step3:选择要比较的两组组(此处查看 group1 与 group2 的物种species_A 丰度是否存在显著差异)
Step4:验证数据是否符合正态分布
Step5:独立样本的 t 检验
Step6:Bonferroni多重检验矫正
结果说明:
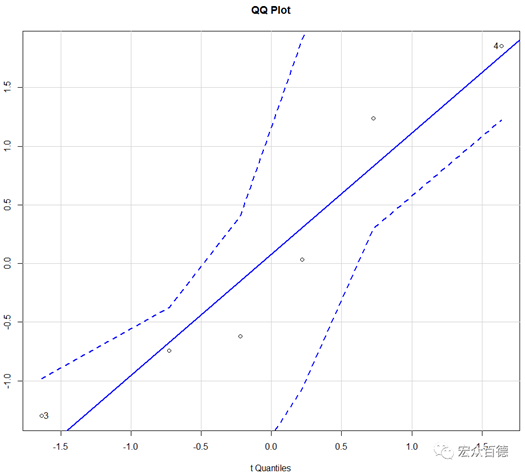
1.正态QQ图:若所有的点都离直线很近,且落在置信区间内(图中虚线部分,默认展示95%置信区间),即表明符合正态性假设,可进行T检验分析。
2. 多重检验矫正意义和原理:
(1) 当同一个数据集有n次(n>=2)假设检验时,通过多重假设检验校正可以大大减少假阳性概率,多重检验矫正方法包括“holm”, “hochberg”, “hommel”, “bonferroni”, “BH”, “BY”, “fdr”,其中bonferroni为最严格的矫正方法。
(2) Bonferroni校正原理是,如果在同一数据集上同时检验n个独立的假设,那么用于每一个假设的统计显著水平为仅检验一个假设时显著水平的1/n。
以上就是该文献生信分析方法体系的实现流程。
