Microbiome: 环境样品中微生物群落物种丰度的绝对定量方法
目前,在环境微生物群落结构组成的研究中,我们常用方法仍是通过扩增分子标签实现微生物群落的相对定量,然而相对丰度无法反映样本中真实的绝对丰度,甚至相对丰度和绝对丰度会存在巨大反差,因此基于样本相对丰度分析可能导致错误的评估。
在《Absolute quantitation of microbiota abundance in environmental samples》一文中,作者采用人工合成的嵌合体DNA作为内参,可以计算得到土壤这类复杂环境样品中的微生物绝对丰度(包括原核、真核和真菌),有助于实现不同样品间以及跨域间的比较,并且强调了绝对丰度与相对丰度在描述菌群变化方面的差异,且在样本中可同时加入针对多个类群(细菌、真菌、真核生物)的嵌合DNA,一次实现多个类群的绝对定量,可以在生物域水平揭示菌群结构的变化。
文章采用一系列已知和未知数量的微生物DNA 的混样策略,测试了定量宏基因组学的准确性,如下图所示。结果表明,土壤的添加不会强烈干扰合成spikes DNA(原核内参用P指示)的共提取和PCR共扩增,但土壤颗粒可能会吸附根瘤菌,且土壤含有腐殖酸,在PCR过程中可能会干扰聚合酶,同时引入了多样性较高的微生物群落,增加了测序难度。内标法spikes在添加或不添加大量根瘤菌细胞的情况下都可以重复性较好的估算出土壤微生物中16S拷贝数丰度。
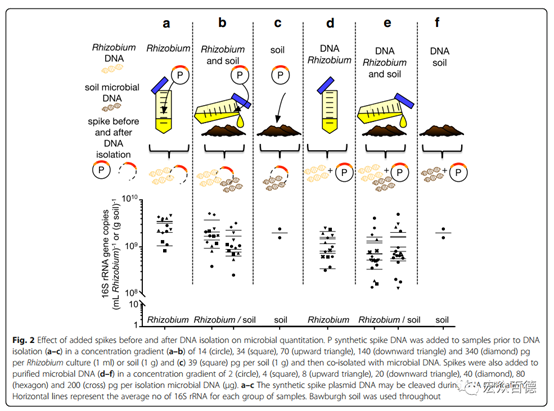
实验数据(Fig.3,实心圆符号)与建模集(Fig.3,线条)分布较相似,表明尽管spikes浓度相差100倍,但仍可以准确估计微生物群丰度。
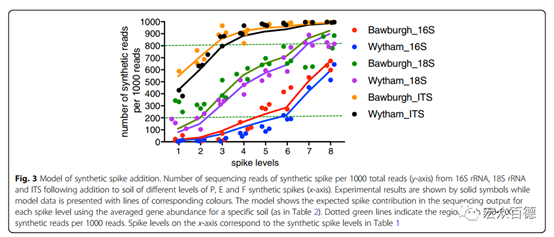
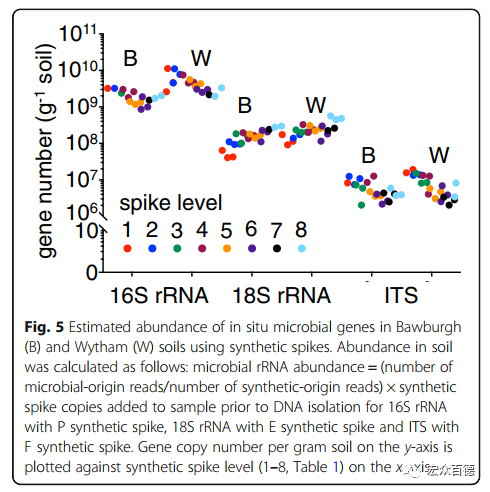
我们观察到基于spikes P和F 值水平添加量的微生物群落丰度估计的梯度值。一般来说,添加的spikes越多,试验结果得到的微生物群落丰度越低。与16S rRNA及其ITS数据集相比,18S rRNA在20–80%比率区内却未观察到这种关系(Fig.5)。这表明,低浓度水平的spikes可能保留在土壤中,因此土壤类型可能导致微生物群估计的显著偏差,而非常高水平的spikes使DNA池过饱和。
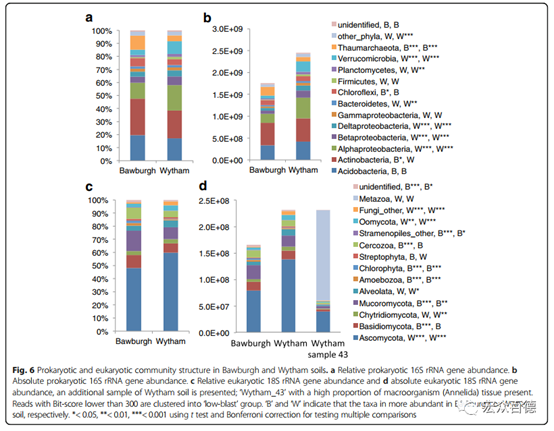
Wytham土壤中原核生物的丰度(约2.5×109)高于Bawburgh土壤(约1.8×109),并比较了微生物群结构的相对丰度(Fig.6a、c)和绝对丰度(Fig.6b、d)。
微生物群落的绝对定量对于微生物生态学研究的各个方面都是必不可少的,该研究方法准确地估计了细菌纯培养样品和土壤样品的微生物数量。添加到土壤中几个数量级的合成spikes的数量与获得的reads序列的数量成正比。活性微生物群的定量将有助于更好地理解环境微生物学中的功能群,并有助于构建更好的微生物群相互作用模型。这种量化分析在与疾病相关的微生物群/亚基因组范围的关联以及土壤生产力相关研究中有着广泛的应用。
该文章提出了一个新的复杂环境微生物群落绝对定量方法,操作相对简单,且可以在样品自身或样品间实现跨域(细菌、真菌等)微生物之间的绝对丰度比较。而且证明在绝对定量过程中,将合成Spikes直接加入到土壤样品中进行共DNA提取、共扩增要比加入到已提取的DNA样品中效果好。另外我们常用的相对丰度表征群落组成变化无法反映样本的真实情况,因此,没有进行绝对定量,微生物群落的潜在生理学或生态学等难以被发掘。
