解读组学图谱帮助石珊瑚应对全球变化
文章链接:
https://www.researchsquare.com/article/rs-4037544/v1
作者:陆祖宏团队(何春鹏、韩婷玉、黄万龙、王冰、廖馨、陈均远、陆祖宏)
文章亮点
1. 完成了全球首批染色体水平高质量石珊瑚基因组的测序和组装。
2. 利用新的工作流程,解决了珊瑚基因组组装中的难点问题,如“外源序列污染”和“高杂合性遗传特征”等。
3. 深化了对石珊瑚基因组保守性和整体演化的认识。
4. 深化了对石珊瑚成钙、共生等广泛关注问题的认识。
5. 讨论了SIDT1等潜在的基因工程工具。
珊瑚礁由于全球气候变化面临前所未有的威胁,包括海温上升、海洋酸化和污染等问题。深入理解石珊瑚多组学对于有效保护珊瑚礁至关重要。然而,高度杂合性和外源序列污染阻碍了高质量石珊瑚基因组的组装。
继2011年第一个石珊瑚Acropora digitifera基因组调查报告发布十几年后,该研究利用新的工作流程和高通量染色体构象捕获(Hi-C)技术,实现了技术突破,成功组装了世界首批染色体水平的高质量石珊瑚基因组。目前,美国国家生物技术信息中心(NCBI)可以搜索到61个石珊瑚基因组,其中11个基于PacBio测序技术达到了染色体组装水平(7个小水螅体石珊瑚和4个大水螅体石珊瑚)。该研究详细介绍了4个组装质量最好、覆盖度最高(至少400倍)的4个小水螅体石珊瑚基因组。其中,A. muricata、Montipora foliosa和M. capricornis的基因组是NCBI的参考基因组,Pocillopora verrucosa的基因组是基于常规评估的最高质量石珊瑚基因组。
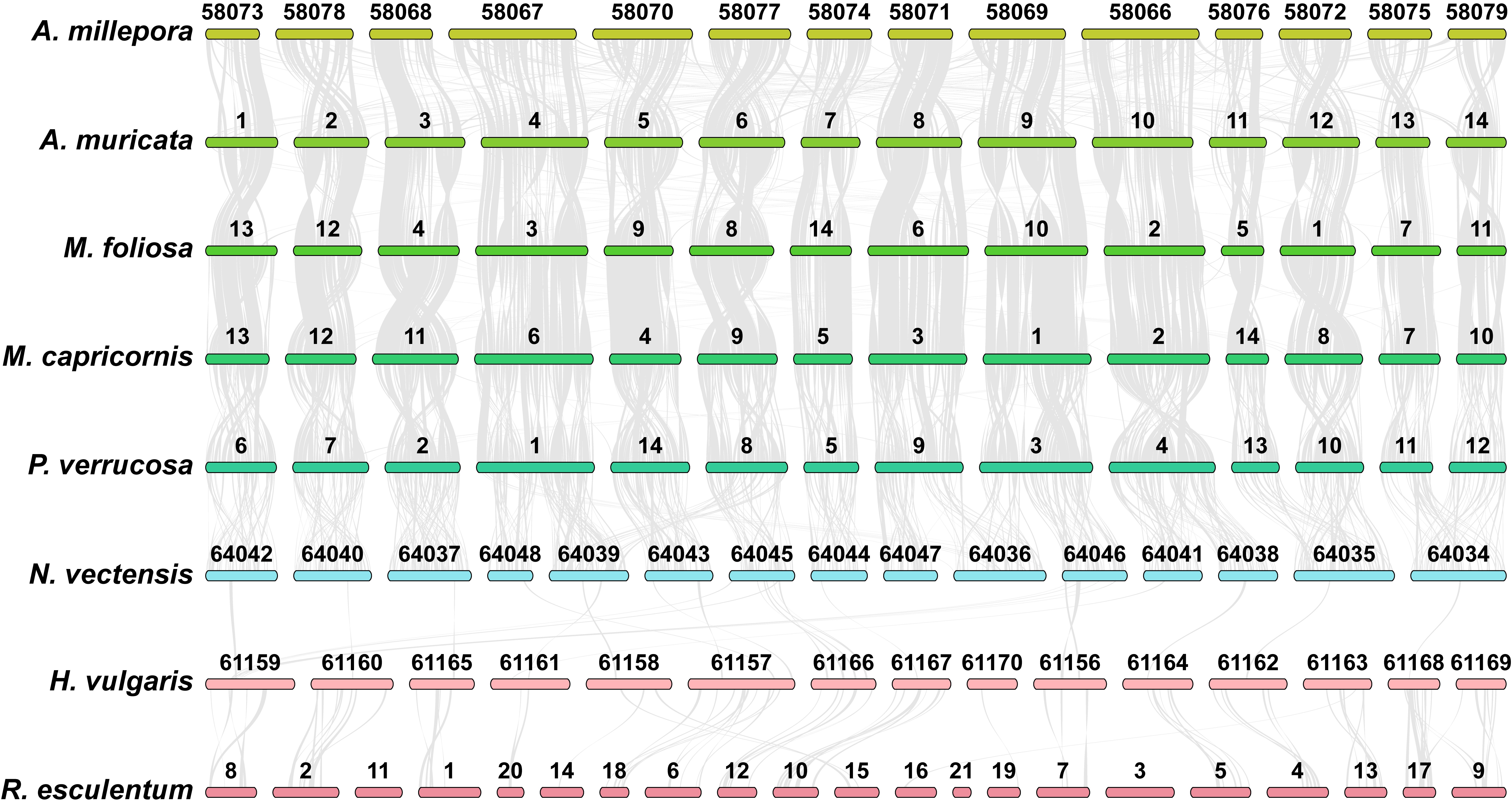
基于这些新挖掘的数据,该研究系统性揭示了石珊瑚的演化历史,通过染色体和同源盒基因的共线性分析,展示了石珊瑚基因组的高度保守性特征。结合单细胞RNA测序(scRNA-seq)和泛基因组分析,深化了对共生、钙化这两个焦点问题的理解。同时,首次展示了(石珊瑚)SIDT1四聚体蛋白跨细胞膜运输miRNA前体的动态模拟过程。
另外,该文关注了最近高频发生的一系列文章撤稿事件。因此,相关分析优先考虑数据及相关发现的可靠性和可复制性,排除了与实际生物学背道而驰的推测性生物信息学分析结果,如石珊瑚与虫黄藻(Symbiodiniaceae)之间的共生调控分析、非模式动物的细胞交流(Cell chat)分析、使用局部选定数据的泛基因组共线性分析、通过筛选scRNA-seq数据风险性挖掘新的亚细胞类型分析,以及基于数字基因家族而非直接序列比对的细胞类型进化分析等。该工作还放弃提及了不能100%重复的具体实验,如利用基因枪和CRISPR/Cas9载体或复合体进行基因编辑试验等。基于scRNA-seq数据和已经被广泛验证的准确可信的已发表文献,该研究详细讨论了共生细胞的三种不同状态和成钙细胞(calicoblasts)的生物矿化过程。此外,出于对同行的尊重,该文避免分析尚未发表、已上传到NCBI的数据。
敬请大家浏览和下载相关论文,提出宝贵意见。
文章链接:
https://www.researchsquare.com/article/rs-4037544/v1
作者:陆祖宏团队(何春鹏、韩婷玉、黄万龙、王冰、廖馨、陈均远、陆祖宏)
相关意见可联系邮箱:zhlu@seu.edu.cn
