微生物群落如何形成? 一个基于功能基因的微生物群落预测模型
2019年北京大学生物医学工程系朱怀球研究团队在Genomics Proteomics Bioinformatics上发表《How Microbes Shape Their Communities? A Microbial Community Model Based on Functional Genes》,我公司对该文章进行深入解读。
文章提出了一个基于功能基因的的微生物群落预测模型(FCP),该模型利用功能基因并考虑环境因素对群落结构的影响进行构建,并提出“群落结构塑造(CSS)基因”是形成特定微生物群落结构的核心功能基因,且证明与所有COGs功能基因或DEG核心基因相比,CSS 基因更能表征群落结构组成。本研究证明不管是针对简单的微生物群落(例如acid mine drainage, AMD)还是复杂的人体肠道微生物群落,FCP预测模型性能均优于广泛的应用的MAP模型。另外,文章指出,在塑造群落结构方面,发挥更重要作用的是少数决定性物种,如古细菌,而非丰度高的物种。
文章主要成果有:
(1)分析AMD微生物群落组成
对17个AMD样品进行层次聚类分析和COG比对分析。
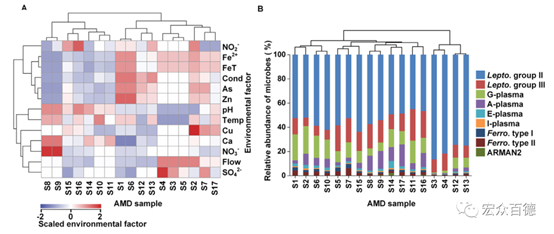
图1 AMD样本的层次聚类分析
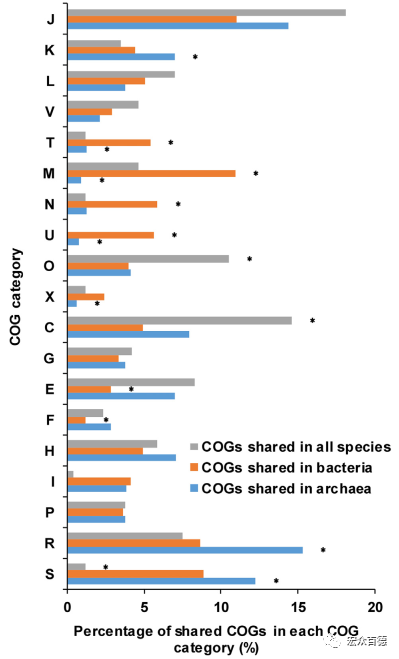
图2 AMD样本中COG分布比较
(2)研究AMD样品中微生物与环境因子的关系
作者用重整置换校正(CCREPE)算法(http://huttenhower.sph.harvard.edu/ccrepe)研究了AMD样品中微生物相对丰度之间的关系。
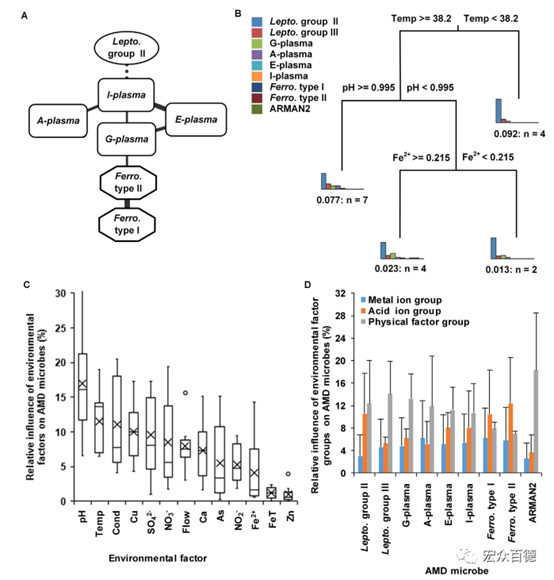
图3 AMD样本中微生物相对丰度与环境因子的关系
(3)预测AMD和人肠道菌群中微生物群落组成
作者用FCP模拟了群落组成对环境因子的响应。
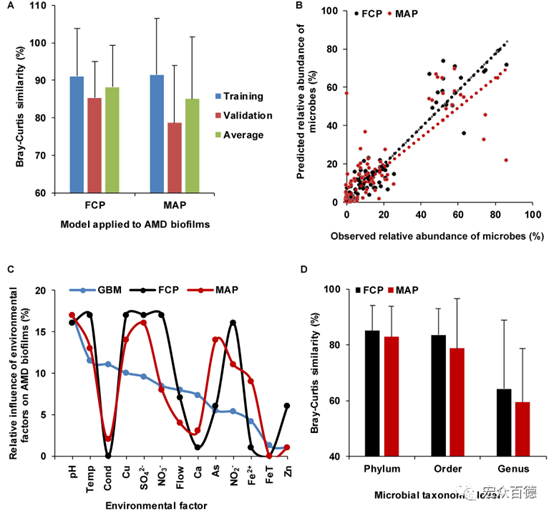
图4 FCP和MAP方法预测精度的比较
(4)鉴定AMD及肠道菌群样本中塑造群落结构的关键CSS基因
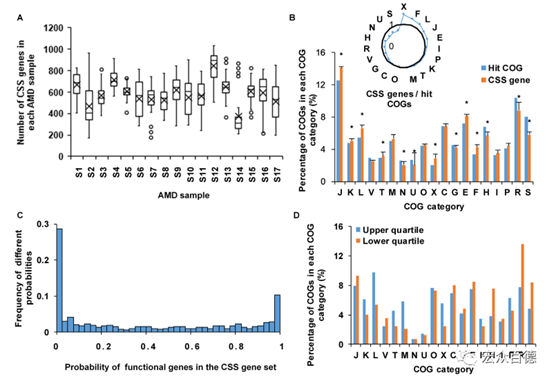
图5 AMD样本的CSS基因分析
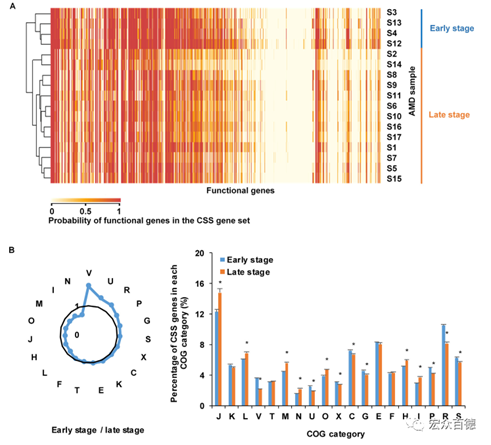
图6早期和晚期演替过程中AMD样品CSS基因的比较
文章提出了一种新的基于功能基因的的微生物群落预测模型,与传统方法相比,该模型更接近群落动态变化且同时考虑了环境因素对群落结构的影响,更能表征群落的真实情况。同时,文章也提出了新概念CSS(Community Structure Shaping)基因,并证明塑造微生物群落结构的基因并非是必需基因,而且群落结构的塑造是功能驱动型的,而非物种。
由于环境微生物群落结构较复杂,影响其组成的因素较多,在建立模型时极易忽略未察觉的关键因子,严重影响预测的准确性。因此需要监测大量的数据不断优化模型,使其鲁棒性更好。另外,文章中物种丰度是由宏基因测序结果得到的相对丰度,是否能反映群落的真实组成,有待考证。
